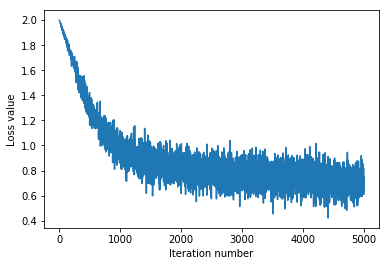
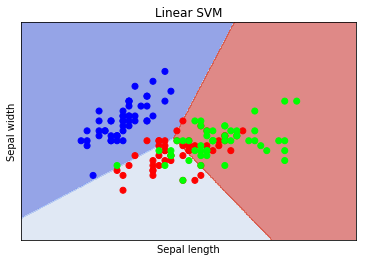
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
| import numpy as np
class LinearSVM(object):
def __init__(self):
self.W = None
def train(self, X, y, learning_rate=1e-3, reg=1e-5, num_iters=100,
batch_size=100, verbose=False):
"""
随机梯度下降法训练
输入:
- X:array(N, D) 训练数据, N 样本数, D 特征维度.
- y:array(N, ) 训练标签, y[i] = c 第 i 个样本的类别标签值
- learning_rate:(float) 学习速率
- reg:(float) 正则化参数
- num_iters:(integer) 迭代次数
- batch_size:(integer) batch size
输出:
- (list) 每次迭代的损失值
"""
num_train, dim = X.shape
num_classes = np.max(y) + 1
if self.W is None:
self.W = 0.001 * np.random.randn(dim, num_classes)
loss_history = []
for it in xrange(num_iters):
idx = np.random.choice(num_train, batch_size, replace=True)
X_batch = X[idx]
y_batch = y[idx]
loss, grad = self.svm_loss(X_batch, y_batch, reg)
loss_history.append(loss)
self.W -= grad *learning_rate
return loss_history
def predict(self, X):
"""
使用训练好的模型预测标签
输入:
- X:array(N, D) 训练数据
输出:
- y_pred:array(N, ) 模型预测标签
"""
y_pred = np.zeros(X.shape[1])
scores = X.dot(self.W)
y_pred = scores.argmax(axis=1)
return y_pred
def svm_loss(self, X, y, reg):
"""
计算损失函数和梯度
输入:
- X:array(N, D) 训练数据
- y:array(N, ) 训练标签
- reg:(float) 正则化参数
输出:
- (loss, gradient) 损失值, self.W的梯度.
"""
scores = X.dot(self.W)
correct_class_score = scores[np.arange(y.shape[0]), y].reshape(y.shape[0],1)
margins = np.maximum(0, scores - correct_class_score + 1)
loss = np.sum(margins) - y.shape[0]
loss /= y.shape[0]
loss += 0.5 * reg * np.sum(self.W * self.W)
matrix = np.zeros((y.shape[0], self.W.shape[1]))
matrix[margins > 0] = 1
matrix[range(y.shape[0]), list(y)] = 1 - np.sum(matrix, axis=1)
dW = (X.T).dot(matrix)/y.shape[0] + reg*self.W
return loss, dW
|